
DNA序列分类997.pdf

金启****富来








亲,该文档总共12页,到这已经超出免费预览范围,如果喜欢就直接下载吧~
相关资料

DNA序列分类997.pdf
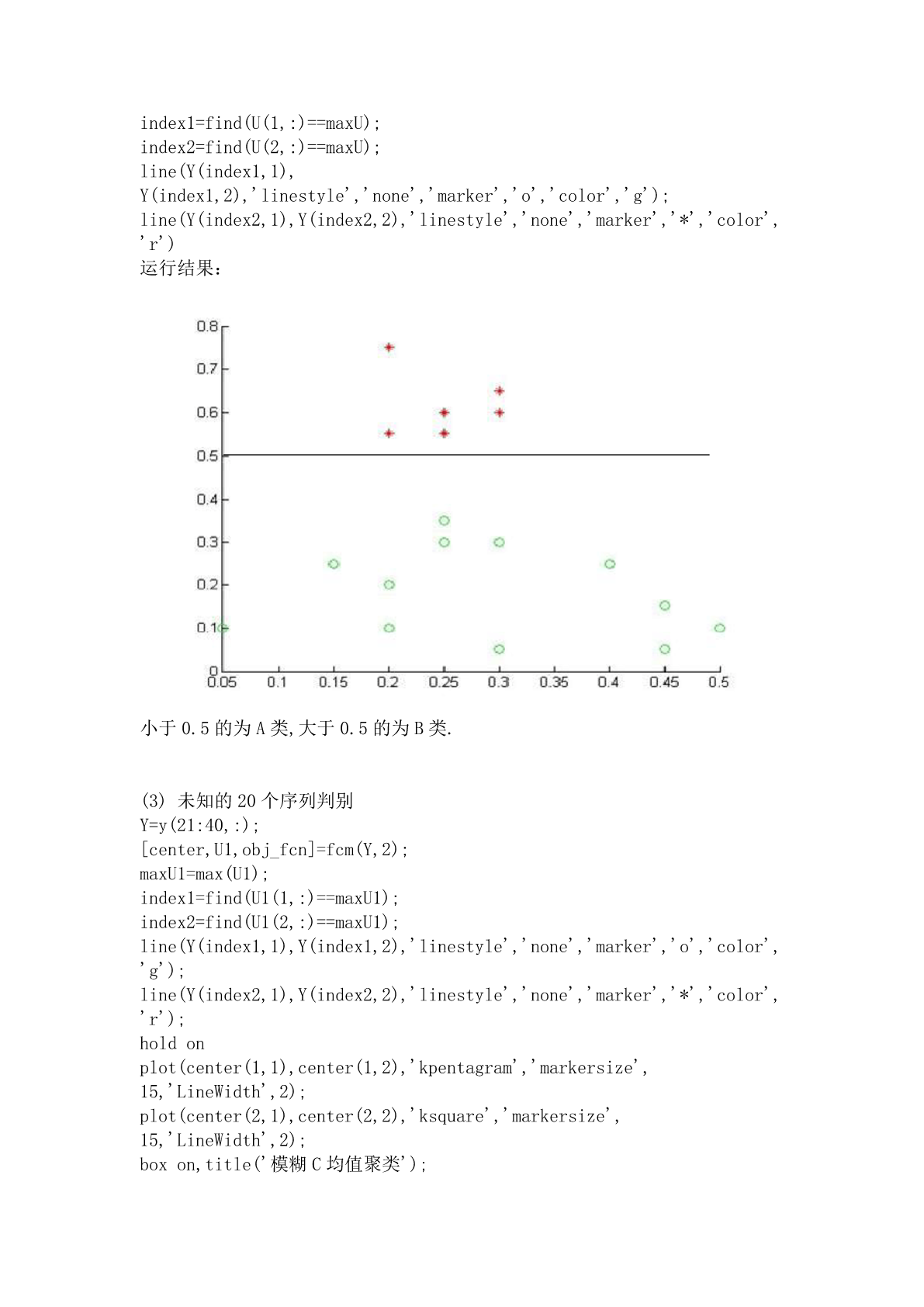
DNA序列分类实验目的学习利用MATLAB提取DNA序列特征建立向量的方法,掌握利用FCM命令进行DNA分类的方法,学会做出分类图形直接给出分类结果的MATLAB编程。知识扩展DNA序列分类DNA(Deoxyribonucleicacid),中文译名为脱氧核苷酸,是染色体的主要化学成分,同时也是基因组成的,有时被称为“遗传微粒”。DNA是一种分子,可组成遗传指令,以引导生物发育与生命机能运作。主要功能是长期性的资讯储存,可比喻为“蓝图”或“食谱”。DNA分子是由两条核苷酸链以互补配对原则所构成的双螺旋结构

DNA序列分类997.doc
DNA序列分类学习利用MATLAB提取DNA序列特征建立向量的方法,掌握利用FCM命令进行DNA分类的方法,学会做出分类图形直接给出分类结果的MATLAB编程。DNA序列分类DNA(Deoxyribonucleicacid),中文译名为脱氧核苷酸,是染色体的主要化学成分,同时也是基因组成的,有时被称为“遗传微粒”。DNA是一种分子,可组成资讯储存,可以引导生物发育与生命机能运作。主要功能是长期性的比喻为“蓝图”或“食谱”。DNA分子是由两条核苷酸链以互补配对原则所构成的双螺旋结合物。其中两条DNA链中对应

DNA序列的分类模型.pdf

DNA序列的分类问题.doc
DNA序列的分类问题摘要本文给出了关于对DNA序列集合如何进行分类问题的解决方案。主要使用了Matlab软件对问题进行建模及运算,运用了聚类分析及判别分析的方法对数据进行处理。在对原始数据的处理和转换方面,使用了C#编写了相关代码,程序完成的主要任务是提取A、T、C、G在各个DNA序列集合中分别出现的频率,然后将这四个频率值作为每个DNA序列集合的特征向量,在Matlab中对其进行聚类分析,并利用分类结果,对未分类的DNA序列集合进行分类预测。1.引言随着人类社会信息和知识总量的不断膨胀,如何有效地管理和

DNA序列分类模型.doc
DNA序列分类模型重庆市数学建模竞赛一等奖王勇,莫志锋,秦力顼(1999级自动化学院)[摘要]本文根据题中所给两个已知类别的DNA序列进行结构特征分析,从中提取信息和构造分类模型,对未知类别的DNA序列进行分类。我们构造了三个分类模型,它们分别是:特征密码子概率分布判别模型、图论最小支撑树模型和向量空间直观判别模型。后两种分类结果几乎一致,判别率在90%左右,误判率控制在(0.05-0.1)范围。问题一结果为:模型一的结果:A类有7个:22,23,27,29,34,35,37;B类有10个:21,24,2
