
基于RBF神经网络的水泥强度预测模型.ppt

xf****65






亲,该文档总共26页,到这已经超出免费预览范围,如果喜欢就直接下载吧~
相关资料

基于RBF神经网络的水泥强度预测模型.ppt
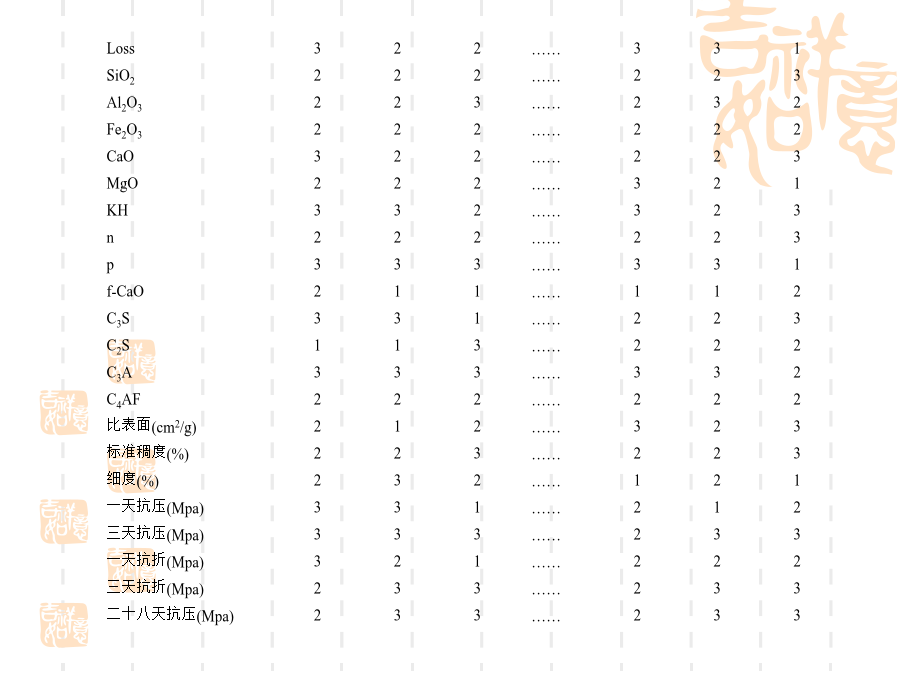
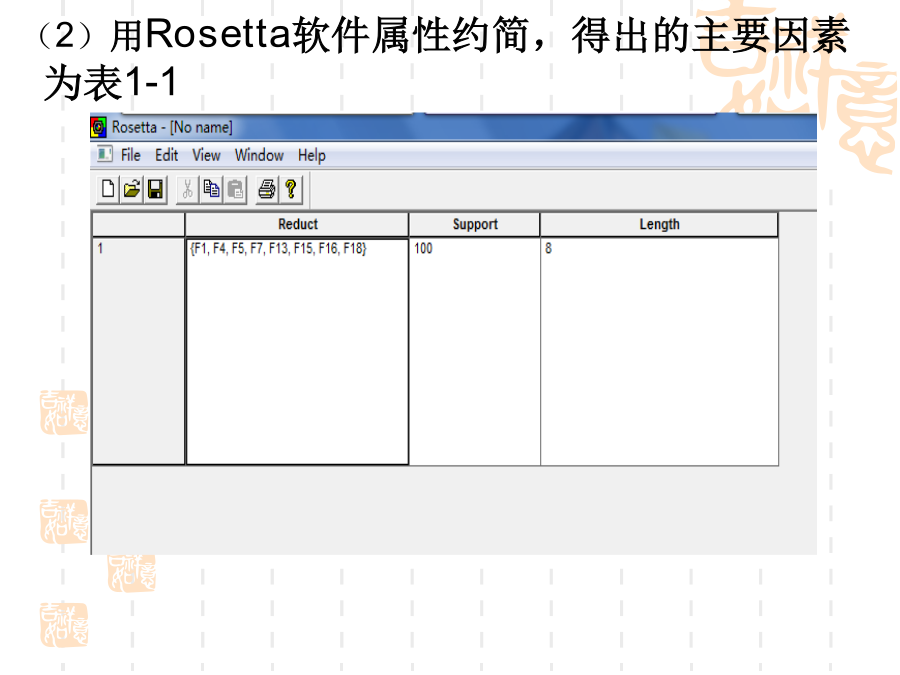
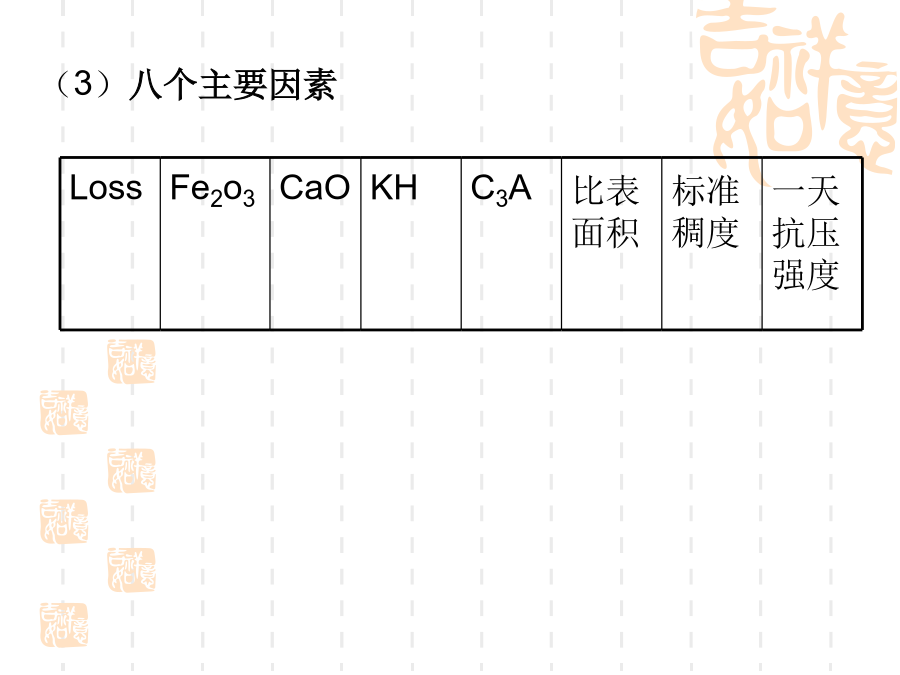
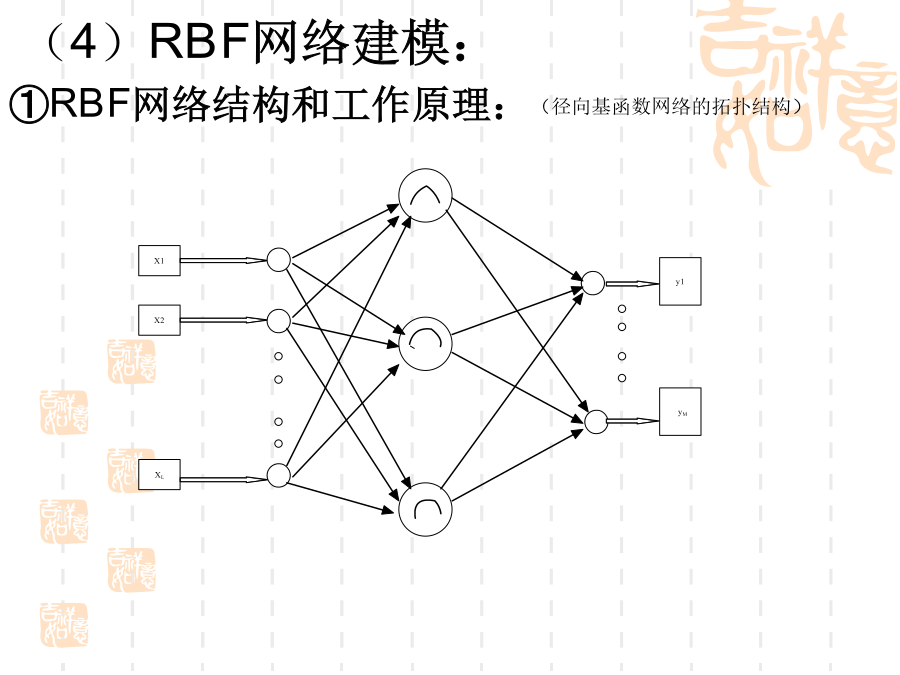
题目:基于RBF神经网络的水泥强度预测模型1.研究内容2.方法及步骤3过程及实现Loss(2)用Rosetta软件属性约简,得出的主要因素为表1-1(3)八个主要因素(4)RBF网络建模:①RBF网络结构和工作原理:(径向基函数网络的拓扑结构)由图对应的公式为:表示输出层第k个节点的输出。表示输出层第k个节点的阈值为径向基函数表示第j个隐层节点连接到输出层第k个节点的权值隐层函数一般采用高斯函数确定:对于高斯函数,其中x是n维输入向量;是第i个函数的中心;是第i个感知的变量(可以自由选择的参数),它决定了

基于RBF神经网络的水质预测模型研究.docx
基于RBF神经网络的水质预测模型研究基于RBF神经网络的水质预测模型研究摘要:在水资源管理和环境保护方面,水质预测是一项重要的任务。准确地预测水质变化可以帮助决策者制定合理的管理策略,以保护水体的健康和可持续利用。神经网络在水质预测中已经得到广泛应用,其中基于径向基函数(RBF)的神经网络模型具有较好的预测性能和计算效率。本论文旨在研究并探讨基于RBF神经网络的水质预测模型,为水资源管理和环境保护提供科学依据。1.引言水是人类生存和发展的基本需求,水质的恶化对环境和人类健康构成严重威胁。因此,准确地预测水

基于RBF神经网络的股票走势预测模型.doc
学士学位论文题目基于RBF神经网络的股票走势预测模型学生殷继平指导教师于延讲师年级2006级专业计算机科学与技术系别计算机科学与技术学院计算机科学与信息工程哈尔滨师范大学2010年5月摘要:股票市场是极其复杂的非线性动力学系统,诸多复杂因素掺杂于其中,使得股市预测异常困难。而神经网络作为非线性动力学系统,具有很强的容错性、自适应性知非线性映射能力,具有可逼近任意非线性连续函数的学习能力和对杂乱信息的综合能力,应用RBF神经网络这种强有力的非线性工具进行研究并进行预测具有着实在的价值、内在的一致性。关键词:

基于RBF神经网络的股票走势预测模型.doc
学士学位论文题目基于RBF神经网络的股票走势预测模型学生殷继平指导教师于延讲师年级2006级专业计算机科学与技术系别计算机科学与技术学院计算机科学与信息工程哈尔滨师范大学2010年5月摘要:股票市场是极其复杂的非线性动力学系统,诸多复杂因素掺杂于其中,使得股市预测异常困难。而神经网络作为非线性动力学系统,具有很强的容错性、自适应性知非线性映射能力,具有可逼近任意非线性连续函数的学习能力和对杂乱信息的综合能力,应用RBF神经网络这种强有力的非线性工具进行研究并进行预测具有着实在的价值、内在的一致性。关键词:

基于RBF神经网络的雾霾成因模型与预测.docx
基于RBF神经网络的雾霾成因模型与预测基于RBF神经网络的雾霾成因模型与预测摘要随着工业化和城市化进程的加快,雾霾问题成为了全球关注的焦点。为了更好地预测和控制雾霾的成因和发展趋势,本文提出了一种基于RBF神经网络的雾霾成因模型。首先,收集了大量的气象数据和环境监测数据,包括温度、湿度、风速、PM2.5浓度等。然后,将这些数据用于训练一个RBF神经网络模型,以预测雾霾的成因以及其发展趋势。实验结果表明,该模型能够很好地预测雾霾的成因和发展趋势,并具有较高的准确性和鲁棒性。本文的研究为雾霾预测和控制提供了新
