
基于多尺度卷积自编码器的脑网络盲源分离方法.pdf

明轩****la







在线预览结束,喜欢就下载吧,查找使用更方便
相关资料

基于多尺度卷积自编码器的脑网络盲源分离方法.pdf
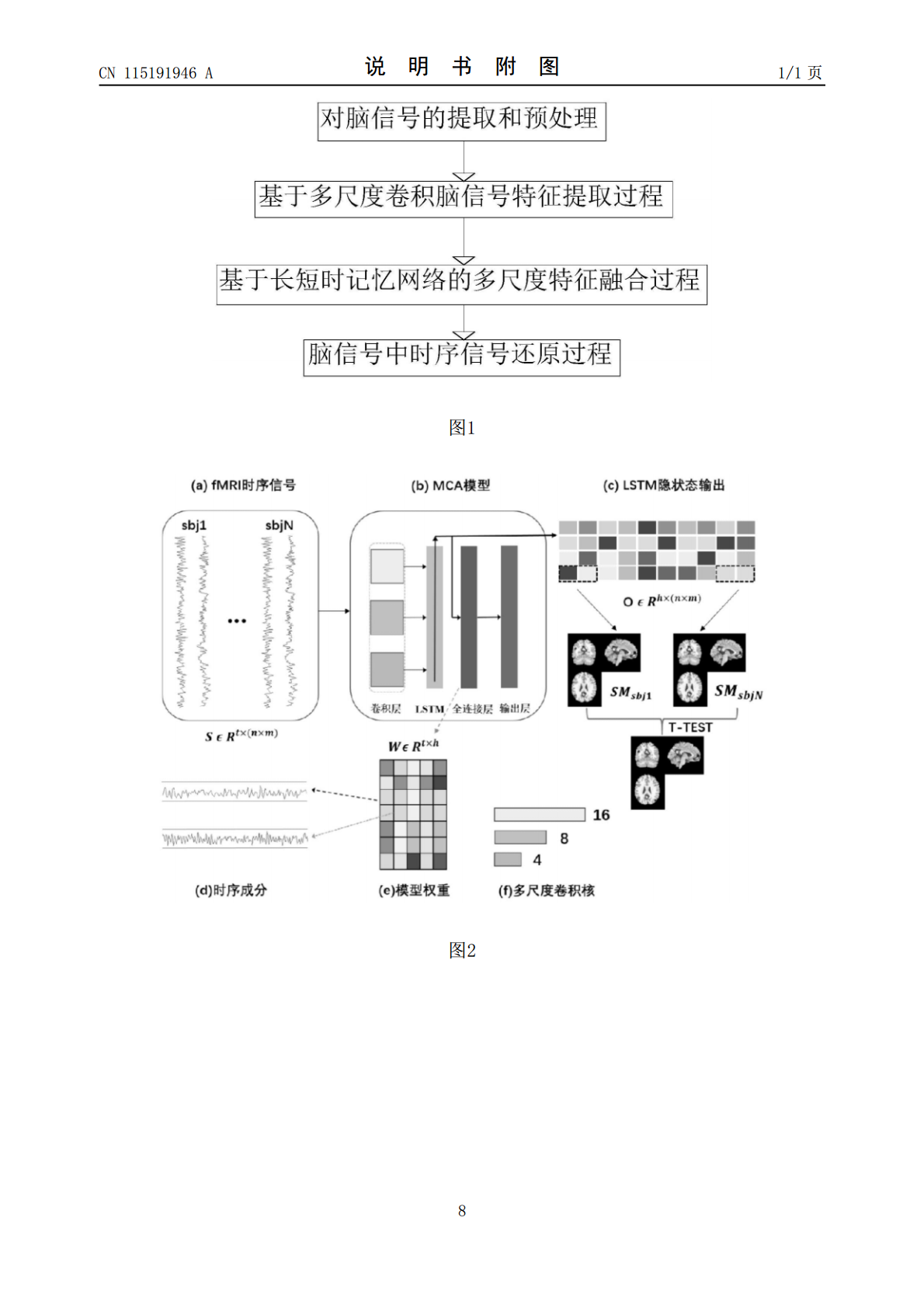
本发明公开了基于多尺度卷积自编码器的脑网络盲源分离方法,涉及脑网络分离技术领域,该基于多尺度卷积自编码器的脑网络盲源分离方法,包括以下步骤:步骤一:对脑信号的提取和预处理;步骤二:基于多尺度卷积脑信号特征提取过程;步骤三:基于长短时记忆网络的多尺度特征融合过程。本发明通过利用一种创新和有效的无监督自编码的学习范式来进行脑网络的盲源分离,与以往的盲源分离技术不同,该模型直接利用可以提取脑信号中不同尺度的时序信息,并利用长短时记忆网络融合各尺度特征,同时对融合后的响应信号进行稀疏限制,从而实现了脑信号中的脑网

基于频点修正的卷积盲源分离方法.pdf
本发明请求保护一种基于频点修正的卷积盲源分离方法,属于信号处理技术领域。通过对源信号短时傅立叶变换(STFT)和相应频点分离信号做特性分析,找出各频点间的相似性;然后,通过设立门限值

一种基于卷积盲源分离的脑电信号独立分量提取方法.pdf
本发明公开了一种基于卷积盲源分离的脑电信号独立分量提取方法,其具体步骤为:搭建包括AD采样模块、短时傅里叶变换模块、频域瞬时盲源分离模块、顺序调整模块和短时傅里叶逆变换模块的基于卷积盲源分离的脑电信号独立分量提取系统;AD采样模块对脑电信号进行采样;短时傅里叶变换模块将时域脑电信号变换到频域;频域瞬时盲源分离模块对频域瞬时混合信号进行分离;顺序调整模块对每个频域段上向量中的独立分量进行顺序调整;短时傅里叶逆变换模块将频域分离结果变换成时域上的独立分量。本发明基于更真实的卷积混合模型提取脑电信号独立分量,采

用于卷积盲源分离的方法和设备.pdf
描述了用于卷积盲源分离的方法和设备。将多个输入信号中的每一个变换至频域中。通过对至少依赖于解混滤波器的系数的代价函数进行梯度下降过程,计算对应于频率区间的解混滤波器的系数的值。在梯度下降过程的每一迭代中,调整用于计算解混滤波器的相同系数的值的梯度项,以改进频率区间上的梯度项的平滑度。关于频率区间中的每一个,通过经由各个解混滤波器对经变换的输入信号进行滤波而估计源信号,其中各个解混滤波器是用系数的计算出的值来配置的。将各个频率区间上的估计的源信号变换至时域。该代价函数适合于评估所估计的源信号之间的去相关性。

频域卷积盲源分离分频段多质心聚类排序方法.pdf
本发明公开了一种频域卷积盲源分离分频段多质心聚类排序方法,(1)将全频带信号进行重叠分组,整个频带被均分为若干个子带,子带之间存在重叠频段;(2)在每个子带内,先后进行多质心聚类和单质心聚类;(3)找出排序失败的频带,对这些频带信号进行重新排序使得该频带处分离信号幅值包络和聚类中心的相关系数之和最大;(4)依次遍历所有子带完成全频带信号的排序。本发明利用重叠子带多质心聚类方法提高了聚类中心,即参考信号的精度,降低子带之间错排的几率。仿真试验表明,本方法具有很好准确性,并且对于短时傅里叶变换窗长在一定范围内
